在细胞的生命活动中,RNA结合蛋白(RBP)与RNA的互作如同精密的“分子开关",调控着mRNA的稳定性、翻译效率及亚细胞定位等关键过程。无论是植物应对盐碱干旱的逆境适应,还是人类疾病中的基因表达异常,这些生命现象的调控机制均与RBP与RNA的动态互作密切相关。然而,传统技术如RIP-seq、CLIP-seq受限于抗体质量和通量瓶颈,难以高效解析这类互作网络。
蓝景科信RAP-seq技术:突破传统的互作研究方案
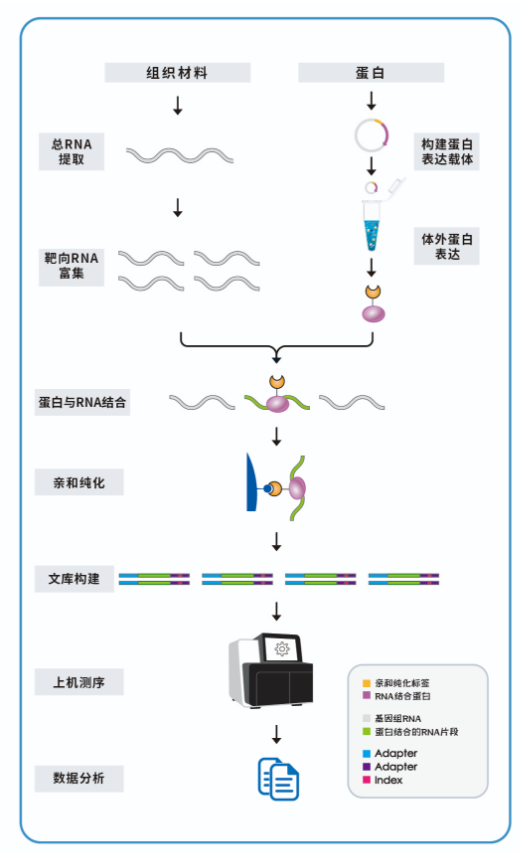
RNA亲和纯化测序(RNA Affinity Purification Sequencing, RAP-seq) 是蓝景科信自主研发的创新技术,通过体外重组蛋白表达-RNA亲和捕获-高通量测序的全流程设计,实现了RBP结合RNA的高效鉴定。
技术流程:
1. 重组蛋白制备:体外表达带标签(如HaloTag)的目标RBP;
2. RNA亲和纯化:重组蛋白与总RNA孵育,特异性捕获互作RNA;
3. 高通量测序和分析:分离结合RNA,构建文库并测序,解析互作转录本。
RAP-seq vs 传统技术:三大优势

RAP-seq的多维应用场景
l 植物逆境应答:解析RBP在干旱、盐碱、高温等胁迫下的调控网络;
l 发育生物学:追踪胚胎发育、器官分化中的RNA-蛋白互作动态;
l 疾病机制研究:筛选癌症、神经退行性疾病中异常表达的RBP及其靶RNA;
l 作物育种:鉴定调控产量、品质的关键RBP,辅助分子育种。
应用案例:RAP-seq在植物逆境研究中的应用
案例一:胡杨耐盐机制的分子解码
文章标题:Populus euphratica GRP2 Interacts with Target mRNAs to Negatively Regulate Salt Tolerance by Interfering with Photosynthesis, Na+, and ROS Homeostasis
期刊:International journal of molecular sciences

技术应用:利用RAP-seq鉴定胡杨GRP2蛋白结合的mRNA,发现其通过靶向光合作用相关基因(如PETC、RBCMT)和抗氧化酶基因(SOD[Mn]、CDSP32),抑制耐盐相关通路,揭示GRP2作为负调控因子的分子机制。
案例二:杨树耐旱基因的互作网络解析
文章标题:Rare variations within the serine/arginine-rich splicing factor PtoRSZ21 modulate stomatal size to determine drought tolerance in Populus
期刊:New Phytologist

技术应用:RAP-seq揭示PtoRSZ21蛋白直接结合自噬相关基因PtoATG2的mRNA,通过可变剪接调控气孔发育,证明该蛋白通过重塑RNA互作网络增强耐旱性。